李川昀课题组发现灵长类RNA编辑的起源新规律
在转录过程中,生物体以DNA为模板,将A、T、G、C四种碱基分别转录为A、U、G、C,从而保证了遗传信息从DNA到RNA的正确传递。然而,在RNA层面,存在一种A-to-I RNA编辑征象,可将RNA分子中的A编辑为I(次黄嘌呤),从而造成DNA与其转录的RNA产物在这些位点的差异。在灵长类中,A-to-I RNA编辑位点数以百万计,且多位于不编码蛋白质的基因组重复区域,因此,探讨这类调控的生物学功能,就成为该领域亟待解决的难题。
近日,北京大学分子医学研究所李川昀课题组通过开展人、猴比较转录组学研究,发现一类特别的A-to-I RNA编辑事件:这些事件仅在特定物种或个体中存在,而在其他物种(或该物种的其他个体)中,响应的DNA位置以核苷酸G的情势存在,因此不具有编辑功能。运用这一特别荟萃,研究发现G-to-A突变位点是A-to-I编辑起源的热点区域,并从理论上证实这些非编码区的RNA编辑具有普遍功能。题为“Evolutionarily significant A-to-I RNA editing events originated through G-to-A mutations in primates”的研究论文在国际着名学术期刊Genome Biology杂志发表。
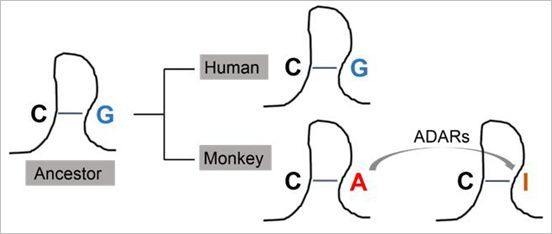
A-to-I RNA编辑事件新近起源自G-to-A突变位点
这类特别的编辑位点,为探讨该调控的生物学功能提供了新角度。一方面,因为这些新调控事件是在G-to-A突变后形成的,通过研究群体中G、A等位基因的分布规律,就可以对该调控的团体功能进行推测。研究注解,这些新近起源的编辑事件受到选择,提醒RNA编辑具有普遍性的生物功能;另一方面,因为这些新近起源的RNA编辑事件在其他近缘物种中是缺失的。因此,探讨RNA编辑功能的题目,就可以转化为对包含或缺失RNA编辑事件的两个物种间进行比较的题目:通过比较这些位点所在基因在表达、剪接、出核等方面的物种间差异,可为揭示RNA编辑事件的普遍功能提供新角度。
李川昀课题组长期运用猴作为人类近缘模式动物的上风,开展基因组学与生物信息学研究。他们邃密解读了猴基因组与转录组(Molecular Biology and Evolution, 2014, 2016),构建了权威的猴基因组知识库RhesusBase(Nucleic Acids Research, 2013),并开展了一系列以猴为角度,探讨人类演化与疾病的特色工作(PNAS, 2018; Nature Communications, 2018; Molecular Biology and Evolution, 2015, 2017; PLOS Genetics, 2012, 2014, 2015)。这些积累为本项工作的顺利完成奠定了基础。
北京大学分子医学研究所博士生安妮、丁晚秋,协和医院杨欣壮博士担任论文共同第一作者,李川昀研究员、陈加余博士担任论文通信作者。本项研究受到国家良好青年科学基金、中组部“万人计划”青年拔尖人才等经费支撑。
编辑:山石
下一篇::生命科学学院魏文胜课题组建立CRISPR混合文库筛选新方法