工学院陈匡时课题组与合作者在解析艾滋病毒增殖机制上取得新进展
近日,工学院生物医学工程系陈匡时课题组和美国芝加哥大学等多所研究机构的科学家运用计算机模仿艾滋病毒(HIV病毒)在分子水平下宿主细胞膜上组装的机制。该研究成果已发表于《美国国家科学院院刊》(PNAS)(链接:http://www.pnas.org/content/early/2017/10/31/1706600114.long)。
Group-specific antigen(Gag)是主导HIV-1病毒组装的核心蛋白,在细胞膜上通过与病毒RNA和蛋白之间交互作用能形成特别很是紧密的病毒复合体,导致膜结构改变,从而使复合体出芽形成病毒颗粒。科学家一向致力于通过各种不同的显微镜手段研究病毒形成的分子机制,但仍然无法在分子层面上明确解析HIV组装过程以及各分子之间的空间和动态举动。Course-Grained Modeling(粗粒化)计算机模仿是一种模仿分子动态举动的手段,可以解析单个Gag蛋白乃至其结构域的构象转变(图1)。通过粗粒化法模仿衣壳(CA)和间隔肽1(SP1)蛋白质结构域(Gag蛋白自组装的紧张成分)、细胞膜和病毒RNA,研究者成功地建立了符合浩繁实验数据效果的病毒颗粒形成模型,还揭示了目前实验手段无法获得的HIV-1可能的组装机制。

图1 CG模仿的CA-SPI构象转变
此项CG模仿研究由芝加哥大学主导,工学院生物医学工程系陈匡时(microRNA克制Gag蛋白组装的发现者)在研究中提供了Gag和病毒RNA交互作用、microRNA克制Gag蛋白组装等在超分辨率显微水平下活细胞中获得的实验数据,对模仿的效果提供了紧张支撑。
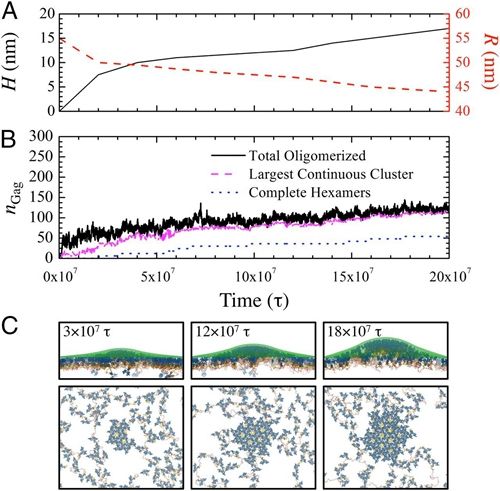
图2 CG模仿的CA-SPI在细胞膜上通过病毒RNA组装的过程
这项研究有望为艾滋病疾病的治疗提供新的思路,并对探讨其他逆转录病毒组装的调控过程具有紧张启示。
编辑:安宁
下一篇::生科院蒋争凡课题组连发两篇文章揭示抗病毒自然免疫旌旗灯号通路中紧张激酶TBK1及IKKαβIKKε的活化机制